- Plateforme
Epitranscriptomique & Séquençage (EpiRNA-Seq)
- Epitranscriptome, ARN, Séquençage à haut-débit, Modifications post-transcriptionnelles

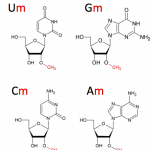
La plateforme Epitranscriptomique et Séquençage (EpiRNA-Seq) est spécialisée et mondialement reconnue dans l'identification et la quantification des nucléotides modifiés au sein des ARN (Epitranscriptomique). Elle s'appuie sur les compétences de l'UMR7365 IMoPA dans le domaine de la biologie moléculaire des ARN.
Installée sur le campus Brabois-Santé de l'Université de Lorraine à Nancy, elle met à disposition de la communauté scientifique académique et industrielle des ressources de haute-technologie (séquenceurs à haut-débit de type Illumina). Depuis 2019, elle est labellisée au niveau local en tant que Structure d'appui à la Recherche (StAR-LUE) dans le cadre du programme INFRA+ de Lorraine Université d'Excellence, et depuis 2021, elle est reconnue au niveau national par le label IBiSA. Elle fait partie du réseau européen COST EPITRAN et organise régulièrement des actions de formation à destination des étudiants et des chercheurs.
La plateforme possède un véritable savoir-faire technique et méthodologique reconnu permettant de contribuer à différents projets d'Epitranscriptomique (RiboMeth-Seq, AlkAniline-Seq, HydraPsi-Seq, RT-Seq) pour aborder des questions scientifiques diverses telles que la dynamique des modifications post-transcriptionnelles dans les ARN.
La plateforme fonctionne principalement en mode collaboratif (implication de la plateforme dans les étapes de votre projet de séquençage de la conception du design expérimental jusqu'à l'analyse statistique des données) mais le mode prestation (principalement séquençage de librairies préparées par vos soins sans implication de la plateforme) est également envisageable dans certaines conditions. La plateforme dispose d'une tarification différentielle (Université de Lorraine, académique et industriel).
La plateforme est ouverte à la communauté scientifique académique et industrielle au niveau local, national et international.
L'équipe est à votre disposition pour vous aider en apportant ses savoir-faires techniques et méthodologiques et vous accompagner de A à Z dans vos projets de séquençage et plus particulièrement d'épitranscriptomique.
La plateforme dispose d'une tarification différentielle (Université de Lorraine, partenaires académiques et industriels).
Tout nouveau collaborateur doit remplir le formulaire de "Demande de prestation" afin de définir la meilleure stratégie à adopter pour la conduite de l’étude, et obtenir un devis. Tout utilisateur s’engagera à accepter les conditions générales (charte utilisateurs). Les instructions pour préparer vos échantillons et les envoyer à la plateforme sont données dans le formulaire "Echantillons".
- Groza P, Kumari K, Esteva-Socias M, Schott J, Bhattarai DP, Sajkowska JJ, Dasgupta R, Peula C, Destefanis E, Williams C, Marchand V, Wikström P, Wiberg R, Campos AB, Gilthorpe JD, Pop B, Mateus A, Motorin Y, Dassi E, Petzold K, Tuorto F, Aguilo F. Fibrillarin-dependent 2'-O-methylation modulates RPS28 ribosome incorporation and oncogenic translation. Cancer Lett. 2025 Nov 17:218124.
 10.1016/j.canlet.2025.218124 ,
10.1016/j.canlet.2025.218124 ,  41260515 ,
41260515 ,  HAL-05376009
HAL-05376009 - Lebret-Kogey V, Ayadi L, Maenner S, Motorin Y, Behm-Ansmant I, Aigueperse C. Quantitative detection of 2'-O-methylated residues in non-coding RNAs using DNAzymes and quantitative RT-PCR. Biochimie. 2025 Oct;237:26-39.
 10.1016/j.biochi.2025.07.006 ,
10.1016/j.biochi.2025.07.006 ,  40633801 ,
40633801 ,  HAL-05162861
HAL-05162861 - Rabany O, Ben Dror S, Arafat M, Aharoni Levitanus H, Halperin Y, Marchand V, Romanovski N, Ussishkin N, Livneh Golany M, Reches A, Wexler J, Mayorek N, Monderer-Rothkoff G, Shifman S, Mâmmer Bouhou W, VanInsberghe M, Pauli C, Müller-Tidow C, Karmi O, Livneh Y, van Oudenaarden A, Motorin Y, Nachmani D. Dynamic rRNA Methylation Regulates Translation in the Hematopoietic System and is Essential for Stem Cell Fitness. Blood. 2025 Nov 5:blood.2024028300.
 10.1182/blood.2024028300 ,
10.1182/blood.2024028300 ,  41191530 ,
41191530 ,  HAL-05353198
HAL-05353198 - Bacci L, Pollutri D, Ripa IJ, D'Andrea M, Marchand V, Motorin Y, Hesse AM, Couté Y, Filipek K, Penzo M. Ribosomal protein L5 (RPL5/uL18) I60V mutation is associated to increased translation and modulates drug sensitivity in T-cell acute lymphoblastic leukemia cells. Biochem Pharmacol. 2025 Oct 31;243(Pt 1):117497.
 10.1016/j.bcp.2025.117497 ,
10.1016/j.bcp.2025.117497 ,  41177179 ,
41177179 ,  HAL-05353080
HAL-05353080 - Hardy L, Marchand V, Bourguignon V, Thuillier Q, Dias C, Krin E, Fruchard L, Yaacov DB, Mazel D, Motorin Y, Baharoglu Z. The tRNA epitranscriptomic landscape and RNA modification enzymes in Vibrio cholerae. PLoS Genet. 2025 Oct 31;21(10):e1011937.
 10.1371/journal.pgen.1011937 ,
10.1371/journal.pgen.1011937 ,  41171884 ,
41171884 ,  Pasteur-05347080
Pasteur-05347080 - International Human RNome Project Consortium. Unlocking the regulatory code of RNA: launching the Human RNome Project. Genome Biol. 2025 Oct 24;26(1):367.
 10.1186/s13059-025-03824-y ,
10.1186/s13059-025-03824-y ,  41137159
41137159 - Belukhina S, Saudemont B, Depardieu F, Lorthios T, P Maviza T, Livenskyi A, Serebryakova M, Aleksandrova M, Ukholkina E, Burmistrova N, Sergiev P, Libiad M, Dubrac S, Barras F, Motorin Y, Marchand V, Hagelueken G, Isaev A, Bikard D, Rouillon C. Specificity and mechanism of tRNA cleavage by the AriB Toprim nuclease of the PARIS bacterial immune system. Philos Trans R Soc Lond B Biol Sci. 2025 Sep 4;380(1934):20240074.
 10.1098/rstb.2024.0074 ,
10.1098/rstb.2024.0074 ,  40904115 ,
40904115 ,  HAL-05353021
HAL-05353021 - Koenig L, Guggenberger V, Eleftheriou K, Pinter Z, Marotto A, Kreutz CR, Wossidlo M, Marchand V, Motorin Y, Schaefer MR. Copy Number Determination of Sperm-Borne Small RNAs Implied in the Intergenerational Inheritance of Metabolic Syndromes. RNA. 2025 May 28:rna.080480.125.
 10.1261/rna.080480.125 ,
10.1261/rna.080480.125 ,  40436631 ,
40436631 ,  HAL-05106950
HAL-05106950 - Leroy E, Challal D, Pelletier S, Goncalves C, Menant A, Marchand V, Jaszczyszyn Y, van Dijk E, Naquin D, Andreani J, Motorin Y, Palancade B, Rougemaille M. A bifunctional snoRNA with separable activities in guiding rRNA 2'-O-methylation and scaffolding gametogenesis effectors. Nat Commun. 2025 Apr 5;16(1):3250.
 10.1038/s41467-025-58664-y ,
10.1038/s41467-025-58664-y ,  40185772 ,
40185772 ,  HAL-05106967
HAL-05106967 - Morin C, Paraqindes H, Van Long FN, Isaac C, Thomas E, Pedri D, Pulido-Vicuna CA, Morel AP, Marchand V, Motorin Y, Carrere M, Auclair J, Attignon V, Pommier RM, Ruiz E, Bourdelais F, Catez F, Durand S, Ferrari A, Viari A, Marine JC, Puisieux A, Diaz JJ, Moyret-Lalle C, Marcel V. Specific modulation of 28S_Um2402 rRNA 2'-O-ribose methylation as a novel epitranscriptomic marker of ZEB1-induced epithelial-mesenchymal transition in different mammary cell contexts. NAR Cancer. 2025 Jan 28;7(1):zcaf001.
 10.1093/narcan/zcaf001 ,
10.1093/narcan/zcaf001 ,  39877292 ,
39877292 ,  HAL-05021643
HAL-05021643

Formation CNRS Entreprises
( -> )
La plateforme EpiRNA-Seq vous propose la formation CNRS Entreprises suivante : "Epitranscriptomics: mapping and analysis of RNA modifications by Next-Generation Sequencing".
Cette formation aura lieu du 17 au 19 mai 2021 sur la plateforme de l'UMS2008/US40 IBSLor au Biopôle à Nancy.
Pour vous inscrire : https://cnrsformation.cnrs.fr/epitranscriptomics-mapping-and-analysis-of-rna-modifications-by-next-generation-sequencing?axe=140

Formation Epitranscriptomique (COST Epitran)
( -> )
La plateforme EpiRNA-Seq organise en collaboration avec I. Motorine (UMR7365 IMoPA) une école thématique Epitranscriptomique avec le soutien du COST Epitran du 10-13 février 2020 à Nancy (France). Cette formation est destinée principalement aux étudiants en thèse et postdocs.